Interattomica
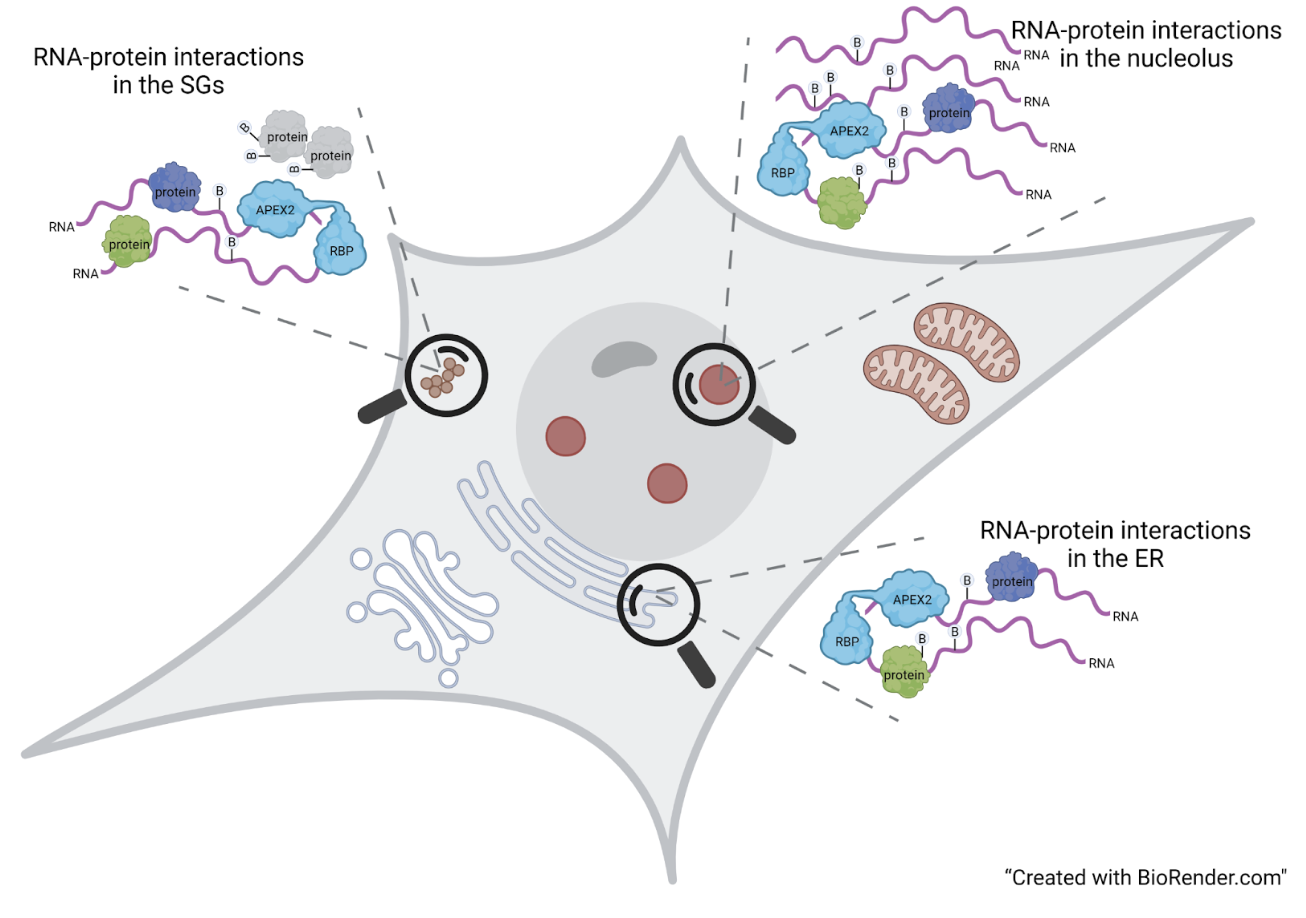
Il laboratorio di interattomica si occupa dell’identificazione delle interazioni RNA-proteina e proteina-proteina all’interno di cellule di mammifero. Queste interazioni sono essenziali per mantenere la corretta omeostasi cellulare e la loro alterazione e’ connessa con numerose malattie umane, incluse malattie neurodegenerative e cancro. Grazie allo sviluppo di sempre piu’ sensibili approcci di RNA-sequencing e Proteomica è possibile oggi identificare gran parte delle interazioni che avvengono nella cellula, incluse interazioni transienti e fra molecole poco abbondanti. il laboratorio utilizza la tecnologia di Proximity Ligation basata sull’attivita’ di specifici enzimi derivati dai batteri che permettono di marcare e purificare sia le proteine che gli RNA presenti in prossimita’ (range < 20 nm) di una proteina di proprio interesse in cellule vive.
In particolare, grazie alla collaborazione attiva con il gruppo del Dr. Francesco Nicassio del Centro di Genomic Science dell’Istituto Italiano di Tecnologia (CGS@SEMM-IIT), il gruppo di Interattomica sta sviluppando degli approcci specifici per l’identificazione di RNA interagenti con varie RNA-binding proteins tramite Nanopore direct-RNA Sequencing.
TEMATICHE DI RICERCA
Tramite l’approccio di RNA-Protein Interaction Detection coupled to Mass Spectrometry (RaPID-MS) (Ramanathan et al., 2017) abbiamo identificato le proteine che interagiscono con i primi e gli ultimi 1.5 Kb del genoma virale che sono le regioni piu’ strutturate e contenenti le regioni regolatorie quali il 5’ ed il 3’ UTR. Questo ci ha permesso di caratterizzare quelle proteine cellulari che interagiscono e potenzialmente regolano la replicazione del SARS-CoV-2 in cellule umane infettate.
STAFF

Roberto Giambruno
Ricercatore